Genee par LGG Hub
Utilities for genetic diagnostic professionals
4 utilisateurs·trices4 utilisateurs·trices
Métadonnées de l’extension
Captures d’écran



À propos de cette extension
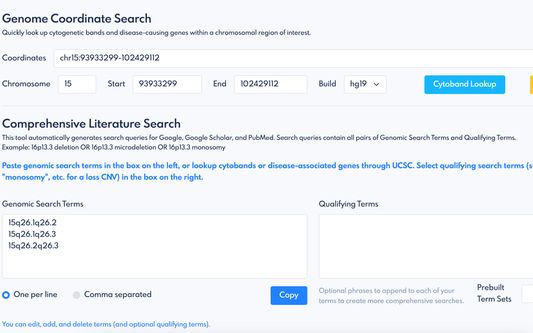
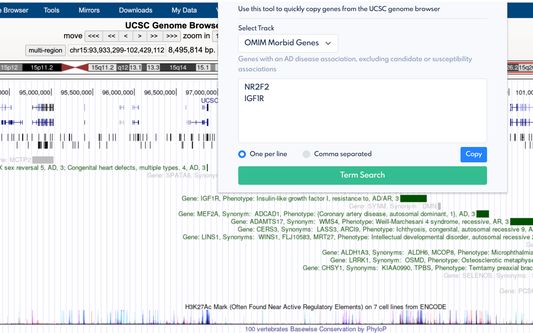
Genee is a browser extension with tools to assist with assessment of genomic regions. Users can input selected genomic coordinates to quickly generate a list of overlapping cytogenetic bands, and use the UCSC genome browser to identify contained OMIM morbid and ClinGen dosage sensitive genes of interest. Cytobands, important genes, or other user-generated search terms for a genomic region can be input and paired with qualifying search terms to describe a type of genomic alteration (e.g. a CNV loss may be described as a deletion, a microdeletion, a loss, a partial monosomy, etc.). The tool quickly generates all pairs of search terms and combines them into search queries that can be used to search for relevant scientific literature via Google, Google Scholar, or PubMed.
Additionally, users of the DECIPHER Genomics Genome Browser can employ this tool to quickly copy all documented phenotypes from any DECIPHER Patient page. The phenotypes can be copied as a comma-separated list or formatted on individual lines.
Additionally, users of the DECIPHER Genomics Genome Browser can employ this tool to quickly copy all documented phenotypes from any DECIPHER Patient page. The phenotypes can be copied as a comma-separated list or formatted on individual lines.
Noté 0 par 1 personne
Autorisations et données
Autorisations nécessaires :
- Ajouter des données dans le presse-papiers
- Accéder aux onglets du navigateur
Autorisations facultatives :
- Accéder à vos données pour les sites du domaine deciphergenomics.org
- Accéder à vos données pour les sites du domaine ucsc.edu
Plus d’informations
- Liens du module
- Version
- 2.1
- Taille
- 1,06 Mo
- Dernière mise à jour
- il y a 3 ans (6 avr. 2023)
- Catégories associées
- Licence
- Tous droits réservés
- Historique des versions
- Ajouter à la collection